高海拔环境的选择压力会驱动生物体表型和遗传的适应。研究组早期的研究表明不同高海拔物种在形态、生理、生化等表型特征出现趋同(Zhu et al. 2018. PNAS),而这种趋同表型的遗传适应机制是多样的,可能受到系统发育背景的严重影响。同时,由于野生鸟类采样困难且转录组测序样品质量要求较高,早期高海拔适应遗传机制的研究多集中在基因序列水平,而以多物种多组织转录调控水平的研究几乎空白(Hao et al. 2019. Current Genomics)。 研究组在同一个系统发育背景下,选择3种来自青藏高原的高海拔雀形目鸟类[褐冠山雀(Lophophanes dichrous)、黑冠山雀(Periparus rubidiventris)、棕额长尾山雀(Aegithalos iouschistos)]和其各自近缘的低海拔物种[沼泽山雀(Poecile palustris)、黄腹山雀(Pardaliparus venustulus)、红头长尾山雀(A. concinnus)]进行了比较转录组学分析。该研究应用第二代高通量测序技术完成了3对高、低海拔物种28个个体5个组织(心脏、肌肉、肝脏、肺脏、肾脏)共128个样品的深度转录组测序,并比较了高、低海拔物种之间在序列水平和表达水平的差异,从而揭示鸟类如何响应高海拔的环境压力。序列比较分析发现:3种高海拔物种在正选择基因上表现出高度相似性(218个共享的正选择基因 ),而氨基酸替代上的相似性极低(3种高海拔物种只有4个基因中包含相同的氨基酸替代位点),暗示了高海拔适应性趋同主要表现在正选择基因水平而非氨基酸替代水平。基因表达比较分析发现:整个基因集的表达谱呈现出组织特异的表达模式(所有物种样品按照组织聚类);而差异表达基因集和海拔相关的基因集的表达谱则呈现出海拔相关的聚类模式,暗示了高海拔环境可能驱动了高海拔物种相似的表达改变。此外,还发现3种高海拔物种筛选到的正选择基因与差异表达基因存在非常低的共享率(2.3%,218个正选择基因中5个基因差异表达),而基因表达、基因连通性及二者和海拔的交互作用与基因的进化速率显著相关。这些结果揭示了3种高海拔鸟类可能以序列和表达水平协同改变的方式实现其适应性进化。 不同于其他类群高海拔适应的研究,该研究首次对野生鸟类开展了多物种、多组织、多尺度的比较分析,拓展了人们对物种如何响应高海拔环境的理解。该项工作以“Comparative transcriptomics of 3 high-altitude passerine birds and their low-altitude relatives”为题于2019年5月24日在线发表于国际权威学术期刊《美国科学院院刊》(Proceeding of the National Academy of Science USA,doi:10.1073/pnas.1819657116 )。中国科学院动物研究所雷富民研究员和屈延华研究员为论文的联合通讯作者;中国科学院动物研究所博士研究生郝艳为论文的第一作者。该项研究得到中国科学院战略先导项目(XDB13020300)以及国家自然科学基金重点、面上项目(31672275、31630069和31572249)的资助。(论文链接) 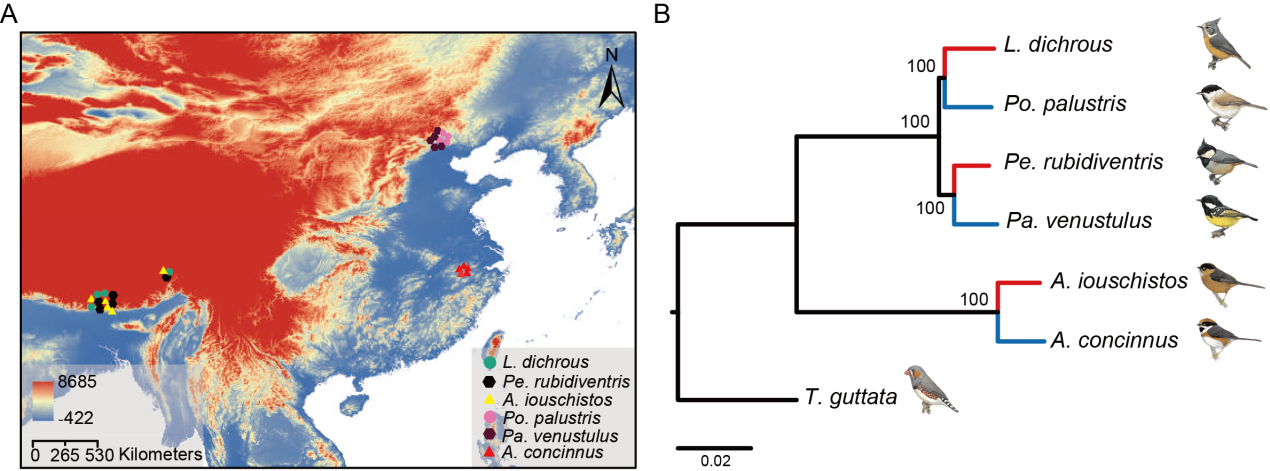
图1.(A)样品采集地 (B)物种系统发育关系 
图2.(A)整个基因集表达谱 (B)整个基因集PCA (C)差异表达基因集表达谱 (D)海拔相关的基因集表达谱 |



